メタゲノムシーケンス
メタゲノミクスでは、環境サンプルからゲノムは、したがって、それは、幅広いアプリケーションで、彼らの自然の生息地で微生物群集を研究するための強力な技術で、前の分離および個々の種の栽培せずに分析し、されています。Novogeneでは、試料中に表さ種、遺伝子、および経路を特定するために、バイオインフォマティクスの専門知識の恩恵を受けながら、微生物群集の豊かな遺伝的レパートリーを探る手助けするNGSにおける当社の専門知識に頼ることができます。
NovogeneはイルミナHiSeqプラットフォームとアセンブリ-最初の戦略とメタゲノム配列決定を提供し、当社のバイオインフォマティクスは、遺伝子予測、機能注釈、分類学上の注釈を提供し分析します。当社の標準的な解析パッケージはMPATH、ヒートマップ、PCA、クラスタ分析およびその他のプログラム、生成する高品質、出版可能なデータが含まれます
サービスの強み
- Highly experienced
- We have completed over 400 metagenomic sequencing projects for our customers, and have published multiple metagenomic studies
- Outstanding service
- We provide high-quality sequencing, an efficient standard workflow, and bioinformatics analyses at a cost-effective price.
- Effective methodology
- Our techniques enhance the generation of data from low-abundance species
- Comprehensive analysis
- Expert bioinformatics analysis with three databases (KEGG, eggNOG, CAZy) provides comprehensive data on annotated genes and metabolic pathways.
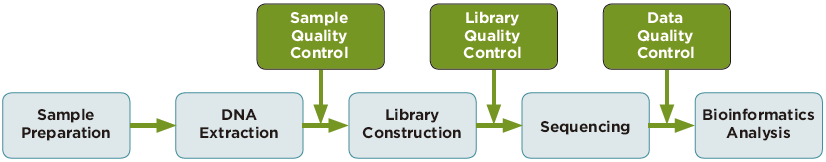
サービスの流れ
- SEQUENCING STRATEGY
- 300-bp insert DNA library
- HiSeq platform, paired-end 150 bp
- DATA QUALITY GUARANTEE
- Each sample generates at least 5-10 Gb raw data, with Q30 ≧ 80%.
- TURNAROUND TIME
- Within 15 working days from verification of sample quality (without data analysis)
- Additional 10 working days for data analysis
- RECOMMENDED SEQUENCING DEPTH
- Two strategies: 5 Gb raw data or 10 Gb raw data
- SAMPLE REQUIREMENTS
- DNA amount: ≧ 1 μg (for one library preparation)
- DNA concentration: ≧ 20 ng/μ
- Purity: OD260/280 = 1.8 - 2.0 without degradation or RNA contamination
- ヒトゲノム
- 全ゲノムシーケンス
- 全エクソームシーケンス
- 遺伝子制御
- RNA-seq(mRNA / LncRNA)
- メチル化DNA解析
- ChIP-seq
- 動植物ゲノム
- de novoシーケンス
- リシーケンス