ChIPシーケンス
ChIP-SEQがヒストン修飾、転写因子、および他のDNA結合タンパク質のためのDNA標的のゲノムワイドなプロファイリングを提供します。それは、回収したDNAのハイスループット配列決定のためのNGSの力で特定のタンパク質-DNA複合体を回収するためのクロマチン免疫沈降(チップ)の選択性を兼ね備えています。タンパク質-DNA複合体は、生細胞から回収されるので、また、結合部位は、あるいは異なる条件下での細胞タイプまたは組織別に比較することができます。
ChIP-Seqでは、濃縮されたDNA領域(タンパク質結合部位)は、バックグラウンド読み込み上記のピークとして検出され、バイオインフォマティクスは、結合モチーフを明らかにすることができ、これらの領域の解析します。アプリケーションは、遺伝子調節、転写複雑なアセンブリ、ヒストン修飾、発達のメカニズム、および疾患過程の研究が含まれています。 NovogenはChIP-Seqのプロジェクト、及びChIP-Seqのための高品質なシーケンシングおよび包括的なバイオインフォマティクス解析を提供することができます
サービスの強み
- Cost-effective
- apid and efficient genome-wide profiling of multiple samples, using only 1/100 of the amount of DNA required for ChIP-chip.
- Comprehensive analysis
- xpert bioinformatics analyses utilizing widely accepted MACS2 software and latest programs for motif prediction, peak annotation, functional analysis and data visualization.
- Professional bioinformatics
- ll PhD team for ChIP-Seq data analysis.
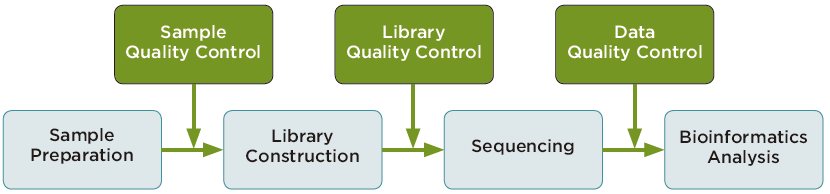
サービスの流れ
- SEQUENCING STRATEGY
- 150~200 bp insert DNA library
- HiSeq platform, single-end 50 bp
- ?150~200 bp insert DNA library?HiSeq platform, single-end 50 bpSAMPLE REQUIREMENTS
- DNA amount ? 50 ng, main peak in 100~500 bp
- TURNAROUND TIME
- 15 working days from verification of sample quality without data analysis
- RECOMMENDED SEQUENCING DEPTH
- ≧ 20 M reads
- 検体要件
- DNA 1.0 μg以上
- FFPE検体から抽出する場合、DNA 1.5 μg以上
- DNA濃度20 ng/μl以上、DNA量10 μl以上。
- OD260/280 = 1.8-2.0、DNAの分解とRNAのコンタミネーションがないこと。
- ヒトゲノム
- 全ゲノムシーケンス
- 全エクソームシーケンス
- 遺伝子制御
- RNA-seq(mRNA / LncRNA)
- メチル化DNA解析
- ChIP-seq
- 動植物ゲノム
- de novoシーケンス
- リシーケンス